Dossier d’Alan dans l’épisode #37.
 Le sujet a été demandé il y a quelques temps par Olivier Tripet et, allez savoir pourquoi, ça m’a tout de suite branché…
Le sujet a été demandé il y a quelques temps par Olivier Tripet et, allez savoir pourquoi, ça m’a tout de suite branché…
Parmi les technologies issues des découvertes scientifiques qui ont impacté en profondeur le fonctionnement de la société, les tests ADN tiennent une belle place puisqu’ils ont bouleversé jusqu’au fonctionnement de la justice elle-même.
L’avantage de l’ADN, c’est que pratiquement tout ce qui est vivant sur terre laisse sa petite trace, clairement identifiable, des bactéries aux baleines en passant par les champignons, les plantes et les différents animaux.
En 1997, un test ADN (test de paternité) post-mortem a révélé qu’Yves Montand, décédé 6 ans plus tôt, n’était pas le père d’Aurore Drossard, qui clamait jusque-là être sa fille cachée.
En 1998, à Seattle, aux Etats-Unis, à l’issue d’une enquête extrêmement compliquée, l’auteur d’un double-meurtre a pu être arrêté grâce aux traces de sang retrouvées sur sa veste. L’analyse ADN indiquait qu’il n’y avait qu’une chance sur 350 millions que le sang retrouvé ne soit pas celui du chien du couple assassiné. Chien assassiné lui aussi d’ailleurs (toute l’histoire ici) . 15 ans plus tôt, cela aurait été complètement impossible, l’assassin serait toujours en liberté.
En 2010, on a pu reconstituer une grande partie de l’arbre généalogique de Toutankhamon, au Caire en partant de restes d’ADN extrêmement fragiles, de plus de 3000 ans.
En janvier 2011, toute la presse du monde se faisait l’écho de la libération de Cornelius Dupree, innocenté au Texas après avoir passé 30 ans derrière les barreaux pour un viol et un braquage qu’il n’avait pas commis. Selon les chiffres d’Innocence Project, une organisation qui se bat pour faire libérer les innocents injustement condamnés, Cornelius est l’un des 271 cas de disculpation aux Etats-Unis depuis l’introduction de la méthode des tests ADN.
Ces quelques exemples suffisent à donner une idée de l’importance et des enjeux des tests ADN et nous allons essayer de les examiner de plus près…
On va commencer par le commencement:
Qu’est-ce que l’ADN?
L’ADN (petit nom de l’acide désoxyribonucléique) est la molécule, présente dans toutes les cellules vivantes, qui renferme l’ensemble des informations nécessaires au développement et au fonctionnement d’un organisme. C’est le support de l’hérédité car il est transmis lors de la reproduction, de manière intégrale ou non. L’ADN porte l’information génétique et constitue le génome des êtres vivants. C’est l’ADN qui va déterminer la couleur des yeux, des cheveux, la stature, la densité des os et de nombreux autres traits. l’ADN est très long (le chromosome 1 humain, tout déplié, mesure 8.3 cm).
Si chaque cellule de notre corps est spécialisée en fonction de son contexte (une cellule de foie ne fait pas le même boulot qu’une cellule de cerveau), leur noyau contient néanmoins un exemplaire complet de l’ADN. C’est vrai de toutes les cellules de notre corps, à quelques exceptions près (les globules rouges du sang par exemple ne contiennent pas d’ADN. Ceci dit, le sang permet tout de même le test ADN grâce aux globules blancs!) Quel que soit le tissu biologique qu’on laisse sur le lieu du crime, il contient de l’ADN, qu’il s’agisse d’un cheveu, d’un poil de nez, de salive, de sang, de sperme. Même de pellicules!
Chaque brin d’ADN est composé de nucléotides. Chaque nucléotide est une combinaison d’acide phosphorique, de sucre (le désoxyribose) et de bases azotées, sortes de lettres de l’alphabet du code génétique. En fait, on s’y réfère comme si c’était des lettres, A pour l’adénine, T pour la thymine, C pour la cytosine et enfin G pour la guanine. L’alphabet de la vie tient en 4 lettres, ça me fascine à chaque fois… On notera que c’est 2 fois plus que le code binaire informatique fait de 1 et de 0, qui permet de faire pas mal de choses lui aussi… Pour décrire une séquence d’ADN en particulier, par exemple un brin contenant 20 nucléotides on pourrait écrire ATTG CCGT ATGT ATTG CGCT.
Ces bases déterminent aussi la complémentarité entre les deux brins de la fameuse double-hélice: quand on a un A à gauche, on a forcément un T à droite et vice-versa. Et en face d’un C, on trouvera toujours un G. On peut facilement déduire un brin à partir de l’autre.
En gros, on peut imaginer l’ADN comme une échelle dont les montants sont faits de sucre-phosphate et les marches de deux bases (AT, TA, CG ou GC) reliées entre elles via un atome d’hydrogène.
La plupart des séquences ADN fournissent les informations permettant aux différentes fonctions de faire ce qu’elles ont à faire. La plupart de ces fonctions, comme les battements du cœur par exemple ou l’échange d’oxygène et de dioxyde de carbone au sein des cellules, sont exactement identiques d’un individu à l’autre. Si vous compariez les quelque 3 milliards de paires de nucléotides de votre génome avec ceux de votre voisin ou avec ceux de la personne la plus différente de vous à l’autre bout de la planète et avec laquelle vous pensez ne rien avoir en commun, vous vous apercevriez que 99.999% de votre ADN est exactement le même!
Les différences d’un individu à l’autre
Alors qu’est-ce qui fait qu’on est si différents les uns des autres et même différents de ses propres parents? L’unicité génétique (c’est à dire le fait que notre ADN soit unique, sauf si on a un vrai jumeau), est le résultat de la reproduction sexuée qui recombine de manière aléatoire le 0.001% des gènes qui nous distinguent les uns des autres à chaque génération. Ces infimes différences sont assez difficiles à traquer. Mais en 1985, une équipe de chercheurs de l’Université de Leicester en Angleterre sous la houlette de Lord Alec Jeffreys, docteur en génétique, met au point une technique pour déterminer une empreinte génétique spécifique à chaque individu (abstract de l’article original publié dans la revue Nature).
Etonnamment, ces empreintes génétiques ne se basent pas sur les 2% des séquences ADN codants, c’est à dire, qui se traduisent en protéines donnant des instructions spécifiques aux cellules mais sur des séquences non-codantes, qui constituent plus de 98% de notre patrimoine génétique et que nous connaissons mieux sous le nom délicat d’ADN poubelle ou “Junk DNA” en anglais car on a longtemps pensé qu’elles ne servaient à rien.)
Chromosomes, locus et allèles…
On va encore examiner quelques généralités génétiques avant d’entrer dans le détail de la méthode des tests ADN afin de bien avoir toutes les cartes en main.
Un chromosome
Les chromosomes sont les supports de l’ADN à proprement parler. L’être humain en compte 46, soit 22 paires homologues (ou autosomes) numérotées de 1-22 et une paire de chromosomes sexuels (XX ou XY). Nous avons donc 23 paires de chromosomes contrairement aux autres hominidés qui en comptent 48 (dans la lignée humaine, 2 chromosomes ont fusionné il y a quelques millions d’années). Le nombre de chromosomes varie grandement d’une espèce à l’autre: le chien (et la poule) en comptent 78, le pigeon 16, le chat 38, la vache 60, le seigle 14, le blé 42, la fougère 1200, le crocus 6… N’essayez pas de trouver une quelconque logique, il n’y en a pas!
Pour bien comprendre comment est organisé notre patrimoine génétique, j’aime bien la métaphore proposée par Matt Ridley dans son livre “The Genome: Autobiography of a Species in 23 Chapters” (2000):
Il y a 23 chapitres appelés CHROMOSOMES. Chaque chapitre contient plusieurs milliers d’histoires appelées GENES. Chaque histoire est faite de paragraphes appelés EXONS, qui sont entrecoupés de publicités, les INTRONS. Chacun de ces paragraphes est constitué de mots de trois lettres, les CODONS. Enfin, chaque mot est écrit avec des lettres, appelées les BASES
Nous avons vu les chapitres/chromosome et les lettres/bases (A, T, C, G). On va garder tout ce qui se trouve entre les deux pour un autre dossier 😉
Un locus
Un locus est un emplacement physique précis et invariable sur un chromosome, comme une adresse sur un échiquier ou un tableur. A la place de A3 ou C6, on trouvera par exemple par exemple 6p21.3. (Exemple piqué à Wikipédia) Le 6 indique la 6e paire de chromosomes; le p indique qu’il se situe sur le bras court du chromosome et le 21.3 révèle la position exacte du locus.
Un allèle
Ce qu’on appelle allèles, ce sont les différentes versions d’un même gène. Là aussi, j’aime bien l’exemple de Wikipedia, très parlant:
Dans le cas du gène déterminant le groupe sanguin A, B ou O, situé sur le chromosome 9 humain (en 9q34.1-q34.2), l’un des allèles, A, code la présence de substance A, un autre, B, la présence de substance B, et un troisième allèle O, ne codant pas d’enzyme actif, détermine, chez l’homozygote O/O, le groupe O.
En d’autres termes, un allèle est l’une des valeurs possibles pour un gène donné, comme une liste déroulante dans un formulaire informatisé.
Les présentations étant faites, passons aux tests ADN à proprement parler…
Les tests ADN
Dans l’ADN non-codant, on trouve des petites séquences qui varient d’une personne à l’autre. On les appelle les STR pour Short Tandem Repeats en anglais (séquences répétées en tandem) ce qui a donné microsatellites en français, ne me demandez pas pourquoi 😉
Une séquence d’ADN-poubelle ressemblera peut-être à ceci (exemple piqué cette fois dans l’excellent “Genetics for Dummies” de Tara Rodden Robinson”
TGCT AGTC AAAG TCTT CGGT TCAT
(sans les espaces évidemment…)
Une courte séquence STR ressemblera plutôt à ceci:
TCAT TCAT TCAT TCAT TCAT TCAT
Le nombre de répétitions trouvées dans des paires de STR sont des allèles et varient d’une personne à l’autre. Dans un même locus, les allèles de la séquence STR ne seront pas les mêmes d’un suspect à l’autre. Ces variations sont appelées polymorphisme (poly = plusieurs, morph = formes). Le polymorphisme naît d’erreurs dans le processus de copie des gènes. Il y a souvent des erreurs de copie, c’est même l’un des moteurs du mécanisme de l’évolution. On appelle ça des mutations génétiques. Lorsqu’elles concernent des fonctions utiles, elles ne passent pas inaperçues: dans la nature, soit elles sont favorables à la survie (comme des dents plus longues par exemple, très utiles en certaines circonstances) soit elles ne le sont pas et se retrouvent éliminées par le mécanisme de la sélection naturelle (dents trop courtes 😉 ). Mais dans l’ADN non-codant, les erreurs restent simplement là et sont passées inaperçues jusqu’aux travaux de l’équipe de chercheurs de Leicester dans les années 1980…
Bref, reprenons notre polymorphisme. Admettons que le chromosome 1 n’ait que deux locus (ou loci). Pour le suspect n°1, par exemple, on pourrait trouver qu’un marqueur STR sur le locus A a la même longueur dans les deux chromosomes (souvenez-vous, à par les chromosomes sexuels chez le mâle (XY), tous les chromosomes viennent en paire). Le suspect n°1 est homozygote pour le locus A.
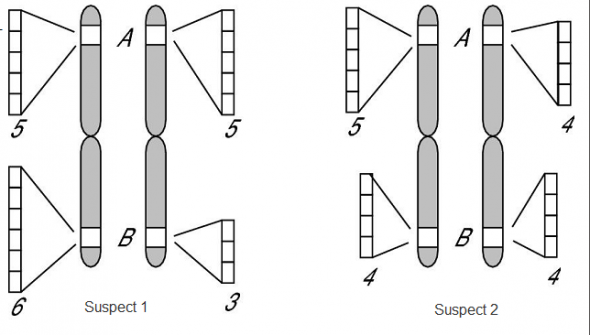
Sur le locus B, le suspect n°1 a deux allèles de longueur différente. Il est hétérozygote pour le locus B.
Prenons maintenant le suspect n°2. Il a des allèles de longueur différentepour le locus A. Contrairement au suspect n° 1, il est hétérozygote pour le locus A (on sait donc déjà à coup sûr que le suspect N°1 et le suspect N°2 ne sont pas la même personne mais ça ne nous dit pas encore qui se trouvait où au moment du crime 😉 ). On regardera quand même le locus B à tout hasard, pour constater que le suspect n°2 a le même nombre de répétitions dans ses deux marqueurs STR et qu’il est homozygote pour le locus B. Tout le contraire de l’autre suspect, quoi…
On a compris le principe. Voyons un peu les détails…
Sur la scène du crime
Sur place, la police scientifique doit commencer par collecter des preuves. Non seulement toutes les traces biologiques humaines (essentiellement sang, salive, sperme, cheveux comme on l’a vu), mais également des traces non-humaines comme les racines, feuilles et pollen des plantes (on va peut-être en retrouver sur les habits d’un suspect, indiquant qu’il se trouvait là…) ou encore des poils ou du sang d’animaux.
Pour collecter leurs échantillons, les enquêteurs doivent être extrêmement prudents: leur propre ADN peut se mélanger comme de rien à l’ADN laissé sur place par les différents protagonistes. Ils doivent porter des gants et des masques, éviter de tousser, d’éternuer. Ils doivent bien sûr couvrir leurs cheveux également (on l’a dit, les pellicules contiennent aussi de l’ADN!)
Ces différentes traces biologiques n’attendent pas suspendues en l’air dans des bulles que les enquêteurs les ramassent… Il faut les chercher partout où c’est possible. Pour la salive par exemple, on prélèvera les chewing-gums, les mégots de cigarettes, les enveloppes, les brosses à dents, rasoirs… Il faut penser à tout. Même les q-tips peuvent révéler des indices! Evidemment, le mieux, c’est de trouver du sang. La plus infime goutte contient quelque 80’000 globules blancs dont le noyau contient un jeu complet d’ADN.
Il faut faire vite
L’ADN, comme toutes les molécules biologiques, peut se dégrader. Une classe particulière d’enzymes, les exonucléases, a pour seul et unique but dans la vie de dévorer l’ADN. Et on en trouve partout bien sûr, sur la peau, sur les surfaces que l’on touche et tout particulièrement chez les bactéries. Les enquêteurs vont donc devoir très rapidement protéger leurs échantillons en les plaçant dans des conteneurs stériles et secs.
De retour au labo
Les échantillons collectés ne contiennent évidemment pas que de l’ADN (ce ne serait pas du jeu 😉 ), la première chose à faire consiste donc à extraire l’ADN des échantillons. Il existe différentes méthodes d’extraction (Anh Tuan l’avait démontré dans l’épisode n°4 de C’est Pas Faux: “Le corollaire de l’ADN”, c’est à 9 minutes 54). En général, on va respecter les étapes suivantes :
- “Ouvrir” les cellules pour libérer l’ADN (ce processus est appelé la lyse. Définition wiki: “On détruit l’intégrité physique de la membrane plasmique des cellules (…) par l’action d’un agent physique, chimique ou biologique (…)”);
- Retirer les protéines, qui constituent l’essentiel de l’échantillon prélevé en les “digérant” avec une enzyme;
- Extraire l’ADN de la solution en y ajoutant de l’alcool
Une fois l’ADN isolé, on l’analyse au moyen d’une technique appelée PCR (pour Polymerase Chain Reaction), ou réaction en chaîne par polymérase en bon français. Aux débuts de la technique, on utilisait une autre méthode, appelée RFLP, mais elle prenait trop de temps et nécessitait des quantités importantes d’ADN pour fonctionner. Selon mes différentes sources, elle n’est plus utilisée aujourd’hui et nous n’allons donc pas en parler.
La méthode PCR
On l’a vu, l’ADN, c’est fragile. N’en avoir qu’un seul exemplaire serait non seulement extrêmement risqué mais de plus, les techniques actuelles ont besoin de disposer de plusieurs exemplaires du même échantillon pour livrer leurs secrets. Le but de la méthode PCR consiste d’abord répliquer à des milliers d’exemplaires le segment spécifique de la molécule d’ADN à étudier. Dans notre cas, des locus – ou loci – STR, afin de constituer l’empreinte génétique.
PCR mode l’emploi:
- Pour répliquer de l’ADN selon la méthode PCR, il faut commencer par séparer les deux brins de la double hélice pour accéder aux bases azotées (les lettres ATCG) normalement protégées par l’acide phosphorique et le sucre. Si on reprend notre image de l’échelle, ce serait comme scier chaque marche au milieu et se retrouver avec deux demi-échelles, dans le sens de la longueur. Ce processus s’appelle la dénaturation. Les liaisons hydrogène qui relient les deux brins sont extrêmement solides mais peuvent être défaites en faisant chauffer la molécule quasi au point d’ébullition (100° C), le reste des composants de la molécule restent intacts. Du coup, on se retrouve avec deux brins dont les bases sont parfaitement accessibles.
- À la fin de la dénaturation, on refroidit la mixture et le processus d’hybridation démarre: dans le double brin, les A d’un brin sont toujours en face d’un T de l’autre brin et vice-versa et les C se mettent avec les G. Chaque brin peut servir de moule à l’autre. Le haut d’un brin doit correspondre au bas de l’autre (en fait on ne parle pas de haut et de bas en génétique mais de polarité 3′ et 5′. L’extrémité 3′ d’un brin se trouve en face de la 5′ de l’autre et vice-versa). Bref, ce qu’on va retenir, c’est qu’avec un seul brin, on peut reconstruire l’autre. Nos brins séparés vont ainsi servir de matrice pour la mise au point d’un nouveau double-brin: on va leur fournir des extraits d’ADN complémentaire appelés “amorces” qu’on aura préalablement marqués avec des teintures fluorescentes, de sorte que les STR de longueur similaire ne puissent pas être confondus les uns avec les autres s’ils proviennent de différents gènes.
- Une fois que les amorces ont trouvé leur segment correspondant, les Taq polymérases entrent en scène. Polymérase, c’est le P dans PCR. Les taq-polymérases sont des enzymes isolées à partir d’une bactérie. Là, on ne va pas entrer dans les détails, on va simplement retenir que ces polymérases vont permettre de construire les nouvelles molécules d’ADN. Cette partie du processus s’appelle “extension”: les polymérases ajoutent des bases azotées à l’extrémité 3′ d’un brin et à l’extrémité 5′ de l’autre en suivant l’ordre de la matrice. À la fin du processus, comme on travaille à plus basse température qu’à l’étape 1, les liaisons hydrogène nouvellement formées sont encore intactes et notre ADN cloné a bien deux brins.
A l’issue de ce premier cycle PCR, on a donc deux copies identiques du segment STR qu’on souhaitait copier. 2 copies ne suffisent pas à la détection de l’empreinte génétique par les lasers. Il faut des centaines de milliers de copies pour que ça fonctionne. Mais comme chaque cycle double le nombre de segments, ça va très vite. Après un deuxième cycle complet dénaturation-hybridation-extension, on a 4 copies. Après 5 cycles, 32 copies. Après 30 cycles: 1’073’741’824 copies du STR cible. Petite remarque: on a fait tout notre calcul comme si on n’étudiait qu’une cellule à la fois. Dans la vraie vie, on parle plutôt de quelques dizaines de milliers de cellules, ce qui donne des billions de copies à l’arrivée 😉
On fait cela avec plusieurs locus (minimum 10 au Royaume-Uni, 13 aux Etats-Unis) et on a notre empreinte génétique!
La lecture des empreintes
Pour cela on utilise un processus appelé électrophorèse. En gros, l’électrophorèse exploite le fait que l’ADN soit chargé négativement: on passe un courant électrique dans un gel et on y injecte l’ADN issu du PCR. L’ADN est attiré vers le pôle positif et les petits fragments STR se déplaçant plus vite que les grands, les STR se trient en fonction de leur grandeur. Et parce qu’on a tagué les fragments avec une teinture fluorescente, un laser piloté par ordinateur est capable de les lire et de les stocker dans l’ordinateur pour les comparer à d’autres empreintes génétiques.
Combien de temps cela prend-il?
L’ensemble du processus n’aura pas duré plus de 24 heures (on est encore loin des délais présentés dans les séries américaines où il suffit d’appeler le labo après 30 secondes pour savoir qui a commis le meurtre, mais c’est déjà pas mal!)
Est-ce fiable?
Il faut garder à l’esprit qu’on parle toujours d’une probabilité. Cette probabilité n’est jamais de 100% (pour cela, il faudrait que l’intégralité des deux génomes comparés soit identique et pas juste quelques locus) et il y a donc toujours un risque d’erreur. À ce stade, je dois faire un nouveau petit crochet dans mon récit. Ça se passe en Angleterre en avril 1999. A l’époque on y réalisait les tests ADN sur 6 locus. La police a retrouvé de l’ADN sur les lieux d’un cambriolage et il se trouve que cet ADN matchait avec l’un des 700’000 échantillons contenus dans la base de données de l’époque. Ni une ni deux, la police se pointe chez le suspect et l’arrête manu-militari. L’homme était malade de Parkinson, à un stade très avancé. Il pouvait à peine s’habiller tout seul et il semblait peu vraisemblable qu’il soit allé jouer les cambrioleurs à 300 kilomètres de chez lui. Mais ce détail n’a frappé personne. Le suspect avait beau clamer son innocence et disposer d’un alibi en béton armé, le test ADN avait parlé. Le risque d’erreur n’était que d’un sur 37 millions. Direction prison. Où il est resté pendant plusieurs mois, jusqu’à ce que son avocat obtienne qu’un nouveau test ADN soit réalisé, sur 10 locus cette fois et pas sur 6. Et bingo… Si les 6 premiers allèles étaient identiques entre l’ADN de l’accusé et l’échantillon trouvé sur place, les 4 suivants en revanche n’avaient rien à voir! C’est depuis ce cas que le Royaume-Uni utilise 10 sites et plus 6 pour ses tests ADN. Les Etats-Unis en utilisent 13, ce qui réduit le risque d’erreur à 1 pour 53 quintillions (si ce mot existe en français?) 53 X 1018, ou 53 suivi de 18 zéros… (53’000’000’000’000’000’000)
Ça, c’est pour les maths. Il y a d’autres sources d’erreurs comme la contamination par exemple. Les virus et les bactéries sont partout et arrivent à se jouer de nos systèmes immunitaires super élaborés… Imaginez le pauvre échantillon d’ADN collecté, sans défense, sans système immunitaire, il peut être très facilement contaminé…
Bref, la méthode est fiable, c’est sans doute ce qu’on a fait de plus fiable depuis l’invention du concept de justice, mais malgré tout, une petite erreur n’est jamais complètement exclue.
Et les tests de paternité?
Deux mots rapidement avant de presque conclure sur les tests de paternité. Chaque chromosome a deux emplacements identiques, l’un provient du père, l’autre de la mère. Pour les tests de paternité, on utilise exactement la même technique qu’on vient d’évoquer mais on ne va simplement pas y chercher les mêmes résultats: si, systématiquement, l’un des deux allèles est identique pour les extraits comparés, il y a un rapport de filiation d’une génération.
Et demain?
Jusqu’ici, les tests étaient en somme une extension de la technique des empreintes digitales: en comparant les traces trouvées sur la scène du crime avec une base de données de traces laissées par des suspects, on pouvait faire le lien entre les deux.
Demain, les tests génétiques pourraient carrément avoir un effet prédictif établissant des portraits-robots sur la base d’un prélèvement ADN. C’est en tout cas l’objectif que s’est fixé le Dr Mark Shriver de l’Université d’Etat de Pennsylvanie qui pense être en mesure de produire la photo de quelqu’un une fois que son équipe aura pu identifier 500 marqueurs faciaux et 500 marqueurs d’hérédité. De ce que j’ai compris, il a encore du boulot: pour le moment, il arrive juste à établir la couleur de la peau, ce qui est un début…
Depuis le début de l’année, une équipe de chercheurs hollando-polonais est capable de prédire la couleur des cheveux de n’importe quelle personne en se basant sur 13 marqueurs présents dans 11 gènes. Le taux de fiabilité de la prédiction est de plus de 90% pour des cheveux roux ou noirs et de 80% pour des cheveux blonds ou châtain. La même équipe avait déjà mis au point l’année dernière une méthode permettant d’estimer l’âge d’une personne à partir de quelques gouttes de sang ainsi qu’une méthode de prédiction de la couleur des yeux.
Ceci dit, l’utilisation de ce genre de techniques est encore de la science fiction aujourd’hui. On n’a pas encore identifié les marqueurs qui déterminent la forme du visage… Et quid de l’influence de l’âge ou de l’environnement sur l’expression des gènes? Les cheveux génétiquement roux ou noirs existent-ils encore? Si oui, sont-ils déjà blancs? Et bon… Les gènes ne nous apprennent rien sur les choix potentiels en termes de shampooings colorants 😉
Voilà… Il y aurait encore eu des milliers de choses à dire, mais je crois que je viens de battre mon record du monde de longueur pour un dossier de Podcast Science. On va donc en rester là pour cette fois. Mais le dossier reste ouvert 🙂
Sources:
- Livres:
- “Genetics for Dummies”, Tara Rodden Robinson, 2005, éditions “For Dummies”http://www.amazon.com/Genetics-Dummies-Tara-Rodden-Robinson/dp/0764595547
- “Genome: The Autobiography of a Species in 23 Chapters”, Matt Ridley, 2000, éditions Harper Perennialhttp://www.amazon.com/Genome-Autobiography-Species-23-Chapters/dp/0060932902
- http://www.scientific.org/tutorials/articles/riley/riley.html
- L’article très complet (comme toujours) de Wikipedia en anglais: http://en.wikipedia.org/wiki/DNA_profiling
- Et l’article en français, pas mal non plus: http://fr.wikipedia.org/wiki/Empreinte_g%C3%A9n%C3%A9tique
- Innoncence Project
- Le cas du chien à Seattle
- http://fr.wikipedia.org/wiki/Test_de_paternité
- La généalogie de Toutankhamon (LeTemps.ch)
- La libération de Cornelius Dupree (RSR.ch)
- l’ADN (Wikipedia)
- Réservé aux geeks: débat sur le nombre d’occurrences de la chaîne “GATTACA” dans les 3’000’000’000 de nucléotides du génome humain 😉 http://boards.straightdope.com/sdmb/showthread.php?t=38598
- L’annonce de la découverte des empreintes génétiques par les chercheurs de Leicester, article original publié dans Nature (abstract): http://www.nature.com/nature/journal/v314/n6006/abs/314067a0.html
- l’ADN-poubelle ou Junk DNA: http://fr.wikipedia.org/wiki/ADN_non_codant
- les chromosomes (Wikipedia): http://fr.wikipedia.org/wiki/Chromosome
- les chromosomes humains (Wikipedia): http://fr.wikipedia.org/wiki/Chromosomes_humains
- la fusion de deux chromosomes dans la lignée humaine: http://www.gate.net/~rwms/hum_ape_chrom.html
- Locus (Wikipedia): http://fr.wikipedia.org/wiki/Locus
- Short Tandem Repeats: http://en.wikipedia.org/wiki/Short_tandem_repeat
- Exonucléases (les enzymes mangeurs d’ADN): http://fr.wikipedia.org/wiki/Exonucl%C3%A9ase
- La lyse: http://fr.wikipedia.org/wiki/Lyse_(biologie)
- La dénaturation: http://fr.wikipedia.org/wiki/D%C3%A9naturation_de_l%27ADN
- Les liaisons hydrogènes: http://fr.wikipedia.org/wiki/Liaison_hydrog%C3%A8ne
- L’hybdridation: http://fr.wikipedia.org/wiki/Hybridation_mol%C3%A9culaire
- Les amorces génétiques: http://fr.wikipedia.org/wiki/Amorce_(g%C3%A9n%C3%A9tique)
- les Taq-polymérases: Taq polymérase
- L’électrophorèse
- L’erreur judiciaire sur la personne atteinte de Parkinson en Angleterre en 1999: http://www.forensic-evidence.com/site/EVID/EL_DNAerror.html
- Portraits-robots basés sur l’ADN:
- http://www.dailymail.co.uk/sciencetech/article-1146503/DNA-left-crime-scene-used-create-picture-criminals-FACE-say-scientists.html
- Papier de l’équipe hollando-polonaise dans Human Genetics: http://www.springerlink.com/content/un82551p2q267g0t/
- Portrait-robot d’après l’ADN: http://www.bbc.co.uk/news/science-environment-12097554
- Prédiction de la couleur des cheveux: http://www.bbc.co.uk/news/science-environment-12111936



