Dossier réalisé par Irène et Claire dans l’épisode 278.
Le prix Nobel de chimie de cette année a été attribué au Français Jean-Pierre Sauvage à l’Ecossais Sir James Fraser Stoddart, et au Néerlandais Bernard Lucas Feringa pour leurs travaux sur la conception et la synthèse de machines moléculaires.
Dans ce podcast bien rodé, Claire et Irène vont voir un peu ce que sont ces machines moléculaires: qu’est ce que c’est, comment ça “marche”, à quoi ça sert, et l’impact de ces connaissances. Notre tandem parfait, cette machine complexe et sophistiquée formée par Claire et Irène, va vous montrer que les machines moléculaires sont bien des machines biologiques, mais que ce sont les chimistes (un peu les physiciens aussi) qui ont permis de mieux les comprendre pour les reproduire et les utiliser en médecine.
Les machines moléculaires : Qu’est ce que c’est?
Tout d’abord, ce sont des machines! Et par machine, on entend un objet capable d’utiliser une source d’énergie pour effectuer par lui-même une ou plusieurs tâches spécifiques, comme une voiture qui transforme l’énergie apportée par l’essence en énergie mécanique pour faire tourner ses roues. Et les machines moléculaires répondent parfaitement à cette définition ! Elles transforment de l’énergie chimique, lumineuse ou encore thermique en énergie mécanique. Autrement dit, elles sont capable d’effectuer un mouvement précis lorsqu’on leur fournit de l’énergie. Dans machine moléculaire, il y a aussi le mot moléculaire, donc ce sont des machines faites d’assemblages de molécules. Ces assemblages ne font que quelques nanomètres (10^-9m) ! Elles sont donc mille fois plus petites que le diamètre d’un de vos cheveux ! C’est grâce à elles que la machine humaine fonctionne, et d’une façon générale tous les être vivants. Cela fait longtemps que les biologistes les étudient et ces machines moléculaires que nos Nobels ont réussit à synthétiser en labo ne sont ainsi pas tout à fait des inventions nouvelles. Mais ils ont bien réussit à en reproduire les mécanismes pour parvenir à en fabriquer et ouvrir ainsi de nouvelles voies en médecine. Alors, nous allons commencer par le début, et voir ce que sont ces machines moléculaires en biologie. Puis Claire va vous expliquer ce qu’il en est de la recherche actuelle en chimie, où l’on reproduit ces machines pour les utiliser notamment en médecine (c’est le but en tout cas).
Les moteurs moléculaires naturels
Prenons tout de suite un exemple simple: vos muscles transforment l’énergie chimique contenue dans les aliments que nous mangeons en énergie mécanique. En se contractant, ils raccourcissent et permettent les mouvements des os. Nos muscles sont en grande partie formés par deux types de filaments qui forment des faisceaux parallèles qui coulissent les uns par rapport aux autres. On a des filaments fins et des filaments épais qui coulissent. La contraction résulte du glissement des myofilaments fins (d’actine) sur des filaments (épais) formés pas de la myosine. Cette molécule de myosine possède des extrémités qui s’attachent sur les filaments. Lorsque de l’énergie chimique est fournie au système (par cassure d’une molécule appelée ATP sous l’action enzymatique de la myosine elle-même mais peut-être aussi grâce à l’énergie thermique), les extrémités se détachent, changent de forme comme le mouvement de nos jambes, et vont aller s’attacher plus loin sur le filament épais. Une vidéo qui simule ce moteur moléculaire:
La deuxième vidéo a été obtenue par un instrument hyper performant, un microscope à super haute résolution, qui fonctionne en milieu liquide sans détruire les échantillons (un AFM). Et n’allez pas croire que les chercheurs sont juste aller positionner le microscope sur les muscles pour prendre une vidéo. Ce serait trop beau. La réalité est bien trop compliquée pour faire cela. Ce que les chercheurs ont fait c’est de mettre dans un petit récipient juste les quelques molécules qui sont nécessaires pour identifier et faire marcher ces machines. Il y a donc une simplification extrême, mais néanmoins le système reste assez proche de la réalité, et que l’on on a enlevé pas mal de paramétres compliqués. Aussi, ne vous attendez pas à une belle vidéo, ce que montre cette vidéo est super difficile à réaliser, on pousse les limites de la technologie à l’extrême, mais regardez si vous pouvez c’est extraordinaire.
Ce que vous voyez c’est cette molécule, attachée par ses deux sortes de pieds à une fibre moléculaire, et qui grâce à de l’énergie chimique va se déplacer le long de la fibre. Un des pieds se détache et avec un mouvement de rotation du reste de la molécule, ce pied va venir se placer devant l’autre pied qui est resté attaché. Et on recommence. En fait, pour être honnête, on ne comprend pas bien encore très bien le mouvement exacte de rotation de la molécule.
Au fur et à mesure que les découvertes en biologie ont permis de les identifier et de comprendre leur fonctionnement, de plus en plus de chercheurs ont cherché aussi à en fabriquer.
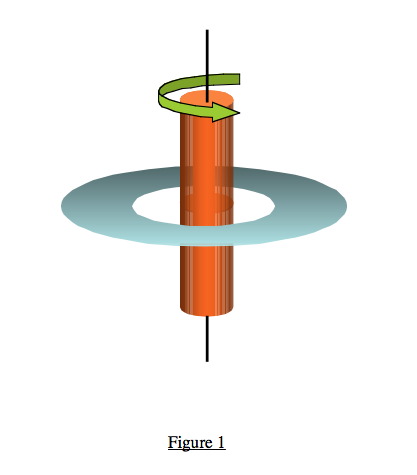
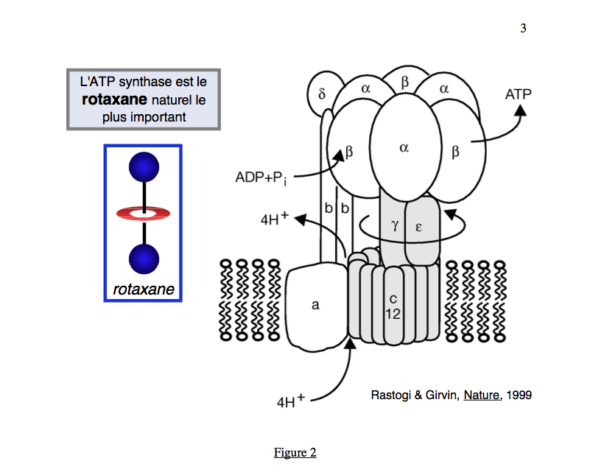
Voyons une description générale de ces petites machines (j’emprunte ici en grande partie un texte écrit par Jean Pierre Sauvage lui-même): Que ce soit en biologie ou en ce qui concerne les machines ou moteurs de synthèse, deux grandes familles de mouvements peuvent être facilement identifiées : mouvement rotatif (Figure 1) ou mouvement linéaire. Dans un moteur rotatif, Il y un axe qui tourne à l’intérieur d’une roue et l’exemple le plus éloquent de cette famille est une enzyme qui s’apelle l’ATP synthétase. C’est une enzyme universelle, découverte dans les années 1990, dont la fonction est de fabriquer l’ATP des organismes vivants, cette molécule riche en énergie qui sert à transporter et transférer cette même énergie. Cette enzyme utilise au départ l’énergie fournie par un gradient de protons H+ (donc de charges électriques) développé de part et d’autre de la membrane sur laquelle elle est fixée. De manière très simplifiée, on peut distinguer un axe, (constitué de l’ensemble γ, ε et c12) et une roue formé par d’autres protéines (figure 2).
Lorsque des protons traversent la membrane, sous la force du gradient électrique, il y a un changement de conformation de l’axe de l’enzyme qui se met à tourner. Après une révolution complète de 360 degrés, il y a formation de 3 molécules d’ATP. Les chercheurs ont pu détecter tout cela en attachant des molécules fluorescentes à la roue qui tourne et en observant les mouvements de molécules fluorescentes. D’une façon plus générale, les moteurs rotatifs sont impliqués dans la synthèse du carburant cellulaire essentiel : l’ATP (adénosine triphosphate) et dans la propulsion de bactéries telles que E. coli De nombreux moteurs linéaires sont également très importants en biologie; Ils participent au transport intracellulaire, à la motilité cellulaire, à la division cellulaire, à l’organisation de la cellule, aux contractions musculaires, aux battements des cils des bactéries. Nous l’avons vu plus haut, un bon exemple est la Myosine, qui permet la contraction musculaire. Voyons en un autre, qui intervient dans la synthèse du code génétique, l’ARN polymérase (Acide Ribonucléique).
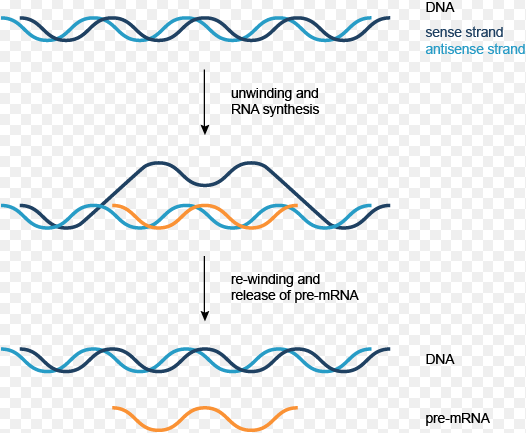
Cette enzyme a été découverte/isolée vers 1960. C’est en fait un complexe constitué par un ensemble de protéines. L’ARN est une molécule qui fait partie du matériel génétique. Elle permet la synthèse des protéines dans nos cellules. Elle est elle même fabriquée à partir de l’ADN (acide désoxyribonucléique). Ces deux molécules ont des structures très proches, elles sont formées par des longs filaments, des brins, et chaque brin est constitué par l’assemblage de petites molécules (les nucléotides) qui s’attachent les unes aux autres, comme les perles d’un collier. Ces perles sont comme les lettres de l’alphabet, selon leur séquence elles forment des mots et les mots vont former des phrases. Donc l’ordre des perles est essentiel, et c’est ce qu’on appelle le code génétique.
En simplifiant un peu, tout d’abord les deux brins de l’ADN se séparent partiellement, et l’ARN polymérase vient s’attacher sur un des brins libres de l’ADN. Ensuite grâce à l’ARN polymérase, tous les ingrédients nécessaires à la synthèse de l’ARN, notamment les perles, viennent se fixer sur l’ARN polymérase elle-même, qui va les attacher les unes aux autres pour former un nouveau brin d’ARN. Simultanément l’enzyme se déplace le long du brin d’ADN matrice, pour lire la séquence des perles. Si elle reconnaît une lettre A, elle attache une lettre A. Puis si la lettre suivante est une lettre C, elle attache une lettre C au nouveau brin qui s’allonge, et ainsi de suite. Finalement elle va rencontrer une séquence qui lui commande de se détacher du brin matrice. Alors elle se détache et l’ADN matrice se referme.
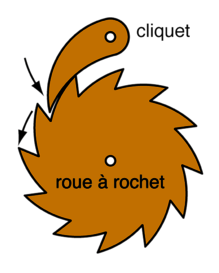
Si on regarde le fonctionnement de cette ARN polymérase, c’est encore plus intriguant. D’abord, comment se fixe-t-elle sur l’ADN, et plus exactement où? L’enzyme reconnaît en fait une séquence bien spécifique de la molécule d’ADN, on appelle cela une séquence promoteur. C’est un enchainement de perles bien spécifique. Il en existe plusieurs selon les espèces, chez nous il y a par exemple ce qu’on appelle la boîte TATA. (Pourquoi TATA, parce que la boîte TATA correspond à une série de 4 lettres/perles: T-A-T-A). Donc l’enzyme reconnaît la séquence T-A-T-A et s’y fixe. Lorsqu’elle se fixe sur la boîte TATA (ou GC), l’ARN polymérase s’associe avec différentes protéines (appelées facteurs de transcription) pour former une particule d’initiation. L’ARN polymérase commence alors sa progression sur l’ADN matrice, en quittant le promoteur. Comment progresse-t-elle le long du brin d’ADN? D’abord, il n’y a qu’un sens possible. Cela fonctionne comme un roue à rochet. Et là il a fallu que je “guouguole” dur pour voir ce que c’est. Et Wiki m’a sauvée: Une roue à rochet est “un dispositif anti-retour limitant un mécanisme rotatif à tourner dans un seul sens. Cette roue est munie sur tout son pourtour d’encoches provoquant dans le sens voulu le soulèvement d’un cliquet pour lui laisser passage mais bloquée par lui dans l’autre sens”.
Quant à l’énergie nécessaire pour faire tourner la roue, est bien apparemment, mais ce n’est pas encore compris du tout, c’est stochastique ie. aléatoire, et probablement il s’agit d’énergie thermique. Autrement dit, il n’y a pas besoin d’un mécanisme bien spécifique qui apporte de l’énergie chimique.
Finalement j’ai choisi aussi de vous parler des hélicases, parce que je les trouve fascinantes et parce qu’on les a identifiées comme responsables de maladies très graves en cas de mauvais fonctionnement. Les hélicases sont des moteurs moléculaires utilisant l’énergie libérée par l’utilisation de l’ATP pour permettre le remodelage des acides nucléiques, et notamment le déroulement de molécules d’ADN double brin. Vous vous souvenez peut-être que les acides nucléiques sont souvent faits de 2 brins et que ces deux brins sont enroulés l’un autour de l’autre pour former une hélice. Pour aller décoder l’ADN, pour permettre à l’ARN polymérase d’aller faire son travail en se fixant sur l’ADN, il faut d’abord que celui-ci soit désenroulé. Donc il y a ces moteurs moléculaires, qui se fixent sur l’hélice d’ADN et enlèvent l’hélice. Leurs mutations peuvent entraîner des troubles du système de réparation avec des conséquences cliniques graves. Parmi les ADN hélicases dont les mutations sont à l’origine de maladies, on trouve notamment des hélicases de la famille RecQ, responsables des maladies génétiques rares comme le syndrome de Bloom par exemple. (le syndrome de Bloom se caractérise par un retard de croissance pré et post-natal ainsi que par des érythèmes survenant le plus souvent au visage lors de l’exposition au soleil. Le diabète et les infections pulmonaires ou auriculaires sont fréquentes. Les garçons sont stériles et les filles présentent une ménopause précoce. Des troubles de l’apprentissage sont habituels. Les individus atteints de ce syndrome ont une incidence de cancer très élevée ce qui constitue la principale cause de mortalité. Ces cancers surviennent précocement). Une autre hélicase est connue pour sa responsabilité en cas de déficience dans l’anémie de Fanconi. D’autres hélicases sont déficientes chez des patients atteints de Xeroderma Pigmentosum (maladie caractérisée par une sensibilité excessive de la peau au soleil, des troubles oculaires et un risque très fortement multiplié de développer un cancer de la peau ou des yeux. Concrètement, les mécanismes de réparation de l’ADN sont inopérants).
En parallèle des découvertes de la biologie au cours des 50 dernières années, la chimie moléculaire a subi une véritable révolution. Les nouvelles méthodes de synthèse, les nouvelles technologies, notamment les nanotechnologies (MEMS pour microelectrochemical systems qui pourraient être utilisés comme des microdétecteurs) ont permis aux chercheurs de concevoir et d’élaborer des systèmes moléculaires de plus en plus complexes, souvent formés de plusieurs constituants. Les chimistes espèrent qu’un jour ils pourront développer des médicaments qui pourront être dirigés très précisément et spécifiquement sur des cellules cibles, comme des cellules cancéreuses par exemple, ou sur le pancréas pour soigner un diabète. Cela permettrait de diminuer les effets secondaires tels qu’on les connaît dans les chimiothérapies. On pense aussi aux micro robots qui pourraient être injectés dans notre sang et qui pourraient aller réparer des tissus endommagés, ou éliminer des cellules cancéreuses.
Un sujet qui me fascine particulièrement: l’utilisation d’ADN pour construire des machines. Il est très facile de nos jours de fabriquer toutes sortes de structures avec des molécules d’ADN. Je mettrai dans les notes d’émission une image prise avec un microscope à force atomique de ce type de structure.

Des chercheurs (du Wyss Institute de l’université de Harvard et Institute of Nanotechnology and Advanced Materials at Bar-Ilan University) sont allés plus loin en fabriquant des nanorobots composés essentiellements de brins d’ADN comme des micro-ordinateurs pré-programmés. Ils ont fabriqué des nanorobots avec de l’ADN formant des petites vésicules contenant des molécules telles que des médicaments et des marqueurs fluorescents. Ils les ont ensuite injectées dans des cafards vivants.
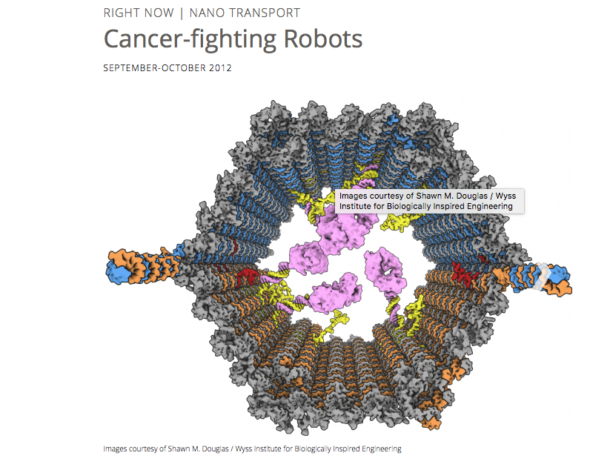
En suivant le trajet des molécules fluorescentes à l’intérieur des cafards, les chercheurs ont pu s’assurer que les molécules avaient bien été délivrées par l’ADN dans les cellules cibles. Il y a en cours des essais cliniques chez des patients (humains donc) atteints de leucémies.
Et c’est donc le moment pour moi de laisser la place à Claire, qui va vous expliquer bien mieux que moi comment les chimistes ont pris (presque) toute la place dans ce domaine… (!).
3) Les machines moléculaires de synthèse
Ce que j’ai prévu de vous raconter c’est comment les chimistes sont arrivés à concevoir et synthétiser des machines moléculaires. Et ce qui est amusant, c’est qu’au départ de cette belle épopée de chimie des machines moléculaires de synthèse qui viennent du coup copier la biologie et qui a été récompensé cette année par un Nobel… et bien il y avait un physicien ! Le génialissime Richard Feynman. Sur lequel Johan a fait un dossier PS ( dossier: 209). Richard Feynman était vraiment visionnaire, puisque dans un discours de 1959 donné à l’American Physical Society, il évoque un domaine de recherche jusqu’alors inexploré : c’est l’infiniment petit : “There’s plenty of room at the bottom”. Il attire l’attention des chercheurs sur la possibilité de construire des machines à l’échelle de la molécule. Une cinquantaine d’année plus tard, challenge réussi pour les 3 Nobel de chimie 2016 !
Comme Irène l’a dit, mais juste pour rappel, ces machines moléculaires répondent à la définition suivante : “un assemblage de plusieurs composés moléculaires distincts, conçu pour effectuer un mouvement en réponse à un stimuli externe approprié”. En d’autres termes, ce sont des assemblages de plusieurs molécules, capables d’effectuer un mouvement de manière contrôlée lorsqu’on leur fournit de l’énergie. Pour moi il y a trois grandes étapes dans la conception de ces machines moléculaires de synthèse : construire un assemblage dans lequel les différentes parties puissent se mouvoir les unes par rapport aux autres, donc dans lequel le mouvement est possible. Puis il faut ensuite réussir à créer le mouvement, puis enfin contrôler ce mouvement via un apport d’énergie.
Première étape donc : construire des assemblages de molécules dans lequel le mouvement est possible.
Et la difficulté qui se pose ici, c’est comment lier ensemble ces molécules dans l’assemblage. Le défi c’est de réussir à créer un lien plus flexible qu’une liaison covalente, qui permette aux différentes parties de l’assemblage de bouger les unes par rapport aux autres. La stratégie adoptée repose sur des chaînes et des noeuds ! Dès les années 60 le défi est lancé par Feynman. Les chimistes vont chercher à produire des chaînes moléculaires. Ce qu’on appelle des chaînes moléculaires en fait ce sont des très longues molécules assez linéaires, un peu comme un fil ou une ficelle. Et ces fils moléculaires, on va vouloir les entremêler. L’idée est que les atomes des différentes chaînes moléculaires ne vont pas interagir entres eux par des liaisons covalentes, mais les chaînes seront néanmoins inséparables les unes des autres parce qu’elles vont être enchevêtrées. Les chaînes peuvent encore bouger un peu les unes par rapport aux autres, mais elles sont confinées ensemble dans l’espace sans aucune interaction entre elles, juste en raison de cet enchevêtrement, ce qui a donné lieu à l’appellation de “liaisons mécaniques” (dont à titre très personnel je ne suis pas spécialement fan, car il n’y a en réalité aucune liaisons). De tels assemblages ont étés synthétisés dès les années 60, comme des caténanes, assemblages moléculaires faits de 2 anneaux imbriqués :
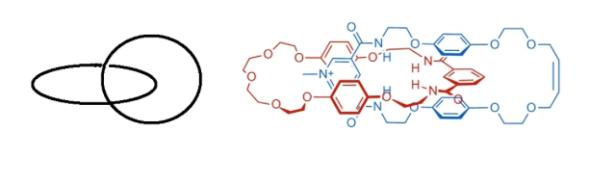
Et aussi des rotaxanes, faits d’un anneau enfilé sur un fil fermé par deux groupements volumineux, qu’on appelle des stoppers, afin d’éviter que l’anneau ne s’échappe.
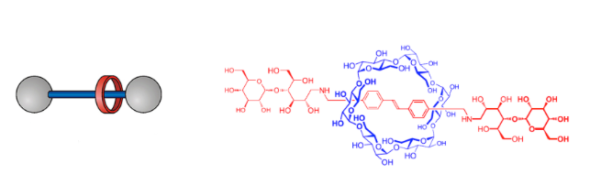
Mais les synthèses avaient un très mauvais rendement, de l’ordre de quelques %. La première vrai avancée a eu lieu en 1983, par le chimiste français Jean-Pierre Sauvage et son équipe. Ils mettent au point un procédé de synthèse des caténanes efficaces, en utilisant un ion cuivre pour un effet template. Un template en chimie c’est un composé, ici l’ion cuivre, qui va organiser la conformation spatiale d’un des réactifs pour faciliter la réaction. Ce n’est pas comme un catalyseur cela ne va pas accélérer la réaction. Mais en arrangeant spatialement un réactif, cela va masquer ou exposer certains de ces sites réactifs et favoriser une conformation finale par rapport à une autre.
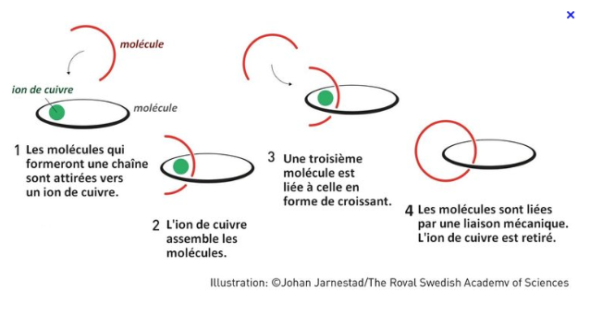
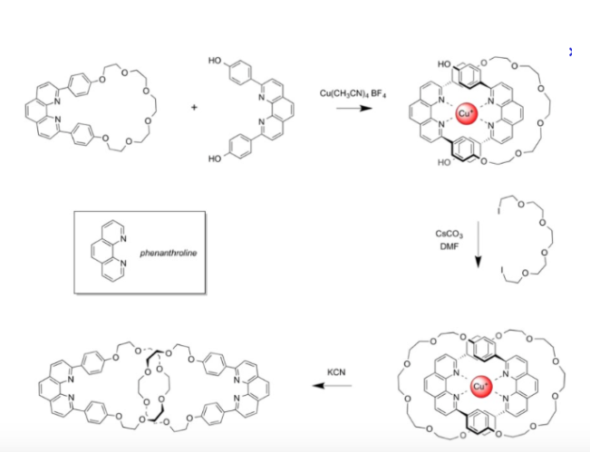
Il y a d’abord coordination avec l’ion cuivre d’une molécule en forme d’anneau et d’une deuxième molécule en forme de croissant. Interactions életrostatiques entre l’ion Cuivre I et les azotes. Une troisième entité en forme de croissant se lie ensuite de façon covalente au premier croissant, formant ainsi le deuxième anneau du caténane. Puis l’élimination de l’ion cuivre permet l’obtention de la structure finale du caténane. Cette technique efficace de synthèse des caténanes, d’un rendement de plus de 40%, est à l’origine d’un regain d’énergie pour ce domaine. Les années qui suivent voient l’apparition de plusieurs très belles structures. Jean-Pierre Sauvage réussit à créer un nœud de trèfle moléculaire, et fut rejoint dans cette voie par James Fraser Stoddart et son équipe, qui ont réalisés de leur côté des anneaux borroméens. De leur collaboration nait un nœud de Salomon. James Fraser Stoddart fut lui-aussi lauréat de ce Nobel, pour la deuxième grande avancée sur la voie des machines moléculaires. Nous sommes en 1991, et l’équipe de Stoddart, qui s’est concentrée sur la synthèse des rotaxanes, rapporte le fait qu’un mouvement de translation de l’anneau le long de l’axe est possible, grâce à des inégalités dans la répartition des électrons. L’anneau, pauvre en électrons, peut se déplacer le long de l’axe vers les zones riches en électrons.
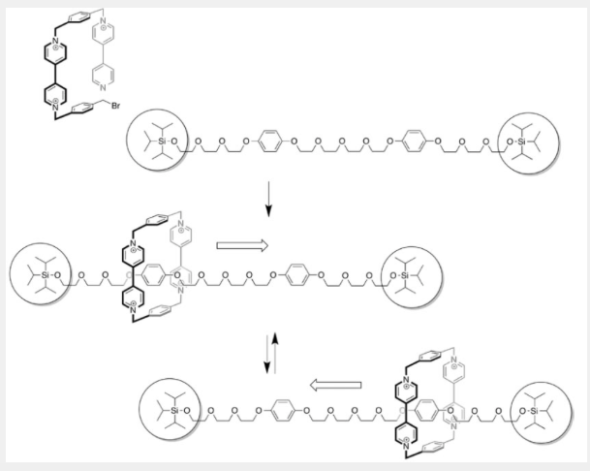
Dans ces deux édifices moléculaires, les différentes parties peuvent se mouvoir : un des anneaux du caténane peut tourner à l’intérieur de l’autre et dans un rotaxane, l’anneau peut glisser le long de l’axe, ou bien avoir un mouvement de rotation autour de l’axe. Mais on était encore loin d’une machine moléculaire : l’enjeu maintenant qu’on sait que le mouvement est possible, c’est de le créer et de le contrôler. Les deux équipes y parviennent en 1994, en introduisant des asymétries dans leurs structures et via un signal électrochimique. L’équipe de Stoddart réussit à faire coulisser de façon contrôlée l’anneau pauvre en électrons entre les deux régions riches en électrons de l’axe de leur rotaxane via un cycle d’oxydation/réduction. De son côté l’équipe de Jean-Pierre Sauvage contrôle le mouvement de rotation dans un caténane, via l’oxydation ou la réduction de l’ion cuivre central. Le contrôle de ces mouvements par un stimuli externe marque un tournant : les premières machines moléculaires de synthèses sont nées ! Et les progrès iront en s’accélérant! En 2000, Jean-Pierre Sauvage met au point un muscle moléculaire, fait de deux rotaxanes entrelacés, qui peut effectuer comme une fibre musculaire un mouvement de contraction/extension.
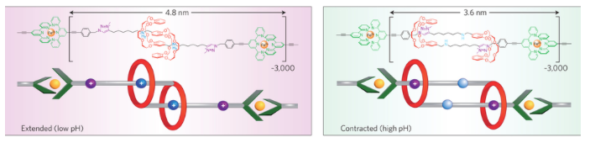
Le groupe de James Fraser Stoddart met au point en 2004 un ascenseur moléculaire, dans lequel une entité peut se déplacer entre deux niveaux séparés de 0,7 nm. En 2007 nait la première puce d’ordinateur moléculaire, dont la capacité de mémoire reste (pour le moment) très modeste. Et Bernard Feringa dans tout cela ? Le troisième lauréat du Nobel et son équipe sont à l’origine du premier moteur moléculaire à mouvement unidirectionnel, mise au point en 1999. Il est difficile de faire aller des molécules dans une seule direction. En réalité, elles s’agitent équitablement dans toutes. Pour empêcher ce caractère aléatoire, l’équipe s’est intéressée au contrôle du mouvement de rotation d’une molécule sur elle-même. L’idée est de lui imposer de tourner toujours dans un même sens. Dans cette approche, les liaisons multiples sont intéressantes, car la rotation autour de celles-ci est bloquée à température ambiante. Un apport d’énergie est nécessaire pour la provoquer, ce qui laisse entrevoir une possibilité de contrôler cette rotation. Leurs travaux ont donné naissance à un assemblage moléculaire constitué de deux molécules planes, semblables aux pales d’un rotor. Les deux pales sont liées entres elles par une double liaison centrale entres deux carbones. Une rotation de 180 degrés s’effectue autour de la double liaison sous l’effet d’un rayonnement UV. Pour empêcher tout retour en arrière, des loquets (en pratique des groupements méthyles) sont attachés à chaque pale. L’impulsion suivante entraîne une rotation dans le même sens, et ainsi de suite. En 2014, le moteur à atteint une vitesse de 12 millions de tours/seconde ! En réponse au défi de Feynman de construire une voiture de taille infinitésimale, Bernard Feringa et son équipe ont construit une « nanovoiture », faite de 4 nanomoteurs jouant le rôle de roue et d’un chassis moléculaire. Challenge réussi haut la main ! Et pas que pour Bernard Feringa ! Puisqu’au printemps à Toulouse va avoir lieu la première course de… nanovoitures ! La “NanoCar Race” se tiendra au Centre d’Elaboration de Matériaux et d’Etudes Structurales (CEMES) du CNRS. La course devait initialement se tenir en octobre, mais elle a été reportée d’un commun accord entre les équipes pour laisser plus de temps de préparation et que le challenge “scientifico-sportif” soit réussi. La course durera 38h, se déroulera à la surface d’un monocristal d’or et les nanovoitures seront pilotées par la pointe d’un microscope à effet tunnel (STM) !
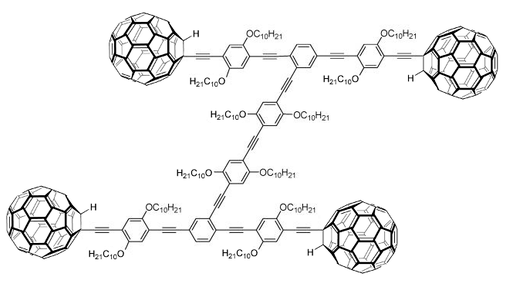
Ici par exemple c’est la première nanovoiture créé par l’équipe du professeur James Tour de l’Université Rice (Texas). Ce sont des fullerènes (C60) qui font office de roues, et la nanovoiture avance par rotation de ces derniers. Pour ceux qui veulent en savoir plus sur cette “NanoCar Race”, je vous conseille l’excellent dossier consacré à cette course dans le numéro 411 de l’Actualité Chimique (octobre 2016). Et maintenant ? En plus d’être de superbes objets chimiques, nul doute que ces machines moléculaires nous promettent des applications spectaculaires, dans la médecine, dans l’informatique et bien d’autres domaines. Contacté à l’annonce de son Nobel de chimie, Bernard Feringa a évoqué le domaine de la médecine. « Imaginez des minuscules robots que les médecins du futur pourront injecter dans nos veines, et qui partiront à la recherche d’une cellule cancéreuse”. Cool non ?
Référence
Actine et myosine: N. Kodera et al. Nature 468, 72 (2010). Kanazawa University
ADN hélicases et maladies associées: lien



